ArrayExpress Gene Expression Atlas
Sokszor felvetődik kérdésként egy génnel kapcsolatban, hogy hol, mikor, milyen körülmények közt, milyen fejlődési stádiumban, minek a hatására, na és milyen mértékben fejeződik ki, expresszálódik. A „klasszikus” molekuláris biológiában ilyenkor például mRNS mintákat izoláltak különböző szövetekből, majd Northern vizsgálattal megállapították, hogy melyik szövetben milyen mértékű az adott gén kifejeződése. A Northern analízis funkcionális genomikai kiterjesztése a micrarray vizsgálat, amely alkalmas arra, hogy a gének (lehetőség szerint az összes gén) közül kiválasszuk azokat, amelyeket keresünk (például egy adott transzkripciós faktor által szabályozottak). Sok-sok ezer ilyen microarray vizsgálatot végeztek el és végeznek szerte a világban. A legtöbbnél kiválasztják az éppen érdekes géneket a saját munkájukhoz, és ezzel párhuzamosan a nyers adatokat beküldik a két nagy microarray adatbázis egyikébe (GEO vagy Arrayexpress). Ezeknek a kísérleteknek van azonban egy érdekes „melléktermékük”, mivel minden kísérletnél elvégzik a „Northern-t” tulajdonképpen az összes génre az adott szövetből, az adott körülmények közt, az adott fejlődési stádiumban az adott indukciós körülmények között. És persze komoly esély van rá, hogy több ezerszer nem pont ugyanolyan körülmények közt végzik a kísérleteket, ezért ha kiveszünk egy gént, akkor megnézhetjük, hogy a különböző sejtekben, szövetekben fejlődési stádiumban stb. mekkora az expressziója.
Ezt persze már korábban is felismerték, mi is használtuk már ezeket az Atlaszokat, amelyek elérhetőek például mindkét microarray adatbázisból. Azonban van egy kis probléma az ilyen összehasonlításoknál, ugyanis ezeket a kísérleteket különböző platformokon, különböző laboratóriumokban, különböző évszakokban és különböző emberek végezték, magyarul nehéz értelmezni az összehasonlításukat. Ezt a problémát kiküszöbölendő az Európai Bioinformatikai Intézet (EBI) Microarray csoportjának kutatói létrehoztak egy Humán Génexpressziós Térképet (Human Gene Expression Map).
A cikk, amelyben leírják az adatbázist, a Nature Biotechnology-ban jelent meg, ami sajnos fizetős (köszönet Szántó Attilának a cikk elküldéséért), de az ábrák többsége a Supplementary Text and Figures fájlban van, ami azonban szabadon letölthető.
Mit csináltak?
- Hogy megszabaduljanak a különböző microarray platformok használata miatti problémáktól az Affimetrix humán U133A csipen végzett 9000 kísérletből kiválasztották az 5372 legmegbízhatóbbat, amelyek 163 különböző laboratóriumból származnak és 206 különböző vizsgálat során keletkeztek.
- A nyers adatokat normalizálták (összehasonlíthatóvá tették), majd a kísérletekhez kapcsolódó leírások és cikkek alapján különböző szempontok alapján csoportosították, például 396 csoportba az szerint, hogy milyen sejt, szövet, betegség stádium, vagy sejtvonalba tartoznak.
- Végső soron kaptak egy 14000 (gének) x 5372 (kísérlet)-es mátrixot amelynek minden pontjához hozzá lehet rendelni néhány más attribútumot vagy dimenziót (például, hogy milyen szövetből származik).
- Ezeket az adatokat aztán különböző algoritmusokkal vizsgálták, legfőképpen PCA (Principal Component Analysis), variancia és klaszter analízissel.
Néhány megjegyzés:
- Az összes matematikai analízist az R csomagot használva végezték (tehát ingyenes programokkal és nagy valószínűség szerint nem windows alatt :-))
- Különböző feltételeket alkalmazva generáltak több mint 40 ábrát.
- A kapott ábrák (matematikai analízisek) eredményeként érdekes biológiai összefüggésekre lehet következtetni, például:
- A PCA alapján három fő tengelyt lehet elkülöníteni, ezek a:
1. Vérképző rendszer, szilárd szövetek és részlegesen differenciált sejt típusok
2. Sejtvonalak, daganatos sejtek és egyéb beteg szövetek
3. Idegszövet
- Az egyik érdekesség, hogy a sejtvonalak együtt csoportosulnak (klasztereződnek) és nem aszerint, hogy milyen szövetből származnak.
- A klaszterezés során egyértelműen a következő típusok különülnek el:
1. Szilárd szövetekből származó sejtvonalak
2. Kötőszövetek és nem teljesen differenciálódott sejtek
3. Normális szilárd és daganatos szövetek
4. Vérképző rendszer
5. Agy
6. Izom és a szív
- Meg lehet vizsgálni, hogy egyes szövettípusoknál / feltételek közt melyek azok a gének, amelyek leginkább változékonyak / eltérő expressziót mutatnak (pl. rákos sejtekre jellemző gének).
- A kapott adatok elég koherensek, de mindazonáltal nem szabad figyelmen kívül hagyni az úgynevezett laborfaktort.
Az adatok használatára / megjelenítésére létrehoztak természetesen egy webfelületet. Sajnos azonban ennek a használhatósága kritikán aluli (igaz még csak béta állapotú, de akkor meg minek kellett már megjelenteni a cikket?). Ez azt jelenti, hogy csak egyszerű keresések vannak, amelyek nehezen használhatók, az eredményekről nincsenek linkek, és nincs semmi segítség, ezér nagyon nehéz értelmezni mind a keresési feltételeket, mind az eredményt. Természetesen egyből kipróbáltam például laborunk egyik legtöbbet vizsgált génjére (PPARg hormonreceptor), és a következő eredményt kaptam (96 olyan csoport a 369-ből, amelyiknél legalább tíz replika áll rendelkezésre).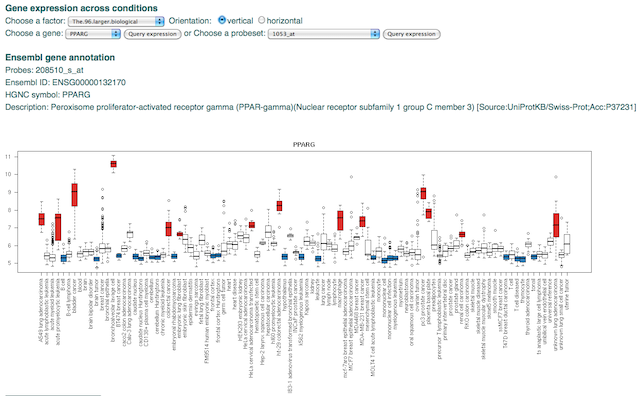




Utolsó kommentek