Az informatika rohamos fejlődése is kevésnek bizonyul olykor a technikai fejlődés kihívásainak leküzdésére (1. ábra). A mindennapi élet és tudomány olyan nagy mennyiségű adatot termelnek, amelynek kezeléséhez, tárolásához, szűréséhez, mozgatásához, feldolgozásához, megjelenítéséhez és például az abban való kereséshez egyre nagyobb kapacitásra van szükség mind az eszközöket mind a munkaerőt tekintve. 2012 óta a világ naponta nagyjából 2.5 exabájt (2.5×260 bájt) adatot generál. A „nagy adat” mérete manapság petabájtos (250 bájt) is lehet, amelynek gyors és kényelmes kezeléséhez a hagyományos módszerek már nem elegendőek (http://en.wikipedia.org/wiki/Big_data). Ilyen „nagy adat”-nak tekinthető a különböző állami és/vagy szolgáltató, például pénzügyi adatbázisok sokasága, illetve a talán még nagyobb ütemben növekvő tudományos adatbázisok. A fizika, biológia és orvostudomány mérései és megfigyelései hasonló adatbázisokba kerülnek, bár az adatok jellege és összetettsége különböző. A meteorológiai és ökológiai megfigyelések és a fizikai modellezések nagyszámú változóval és viszonylag nagy felbontással történnek, míg az idegrendszer kapcsolatait vizsgáló konnektomika hasonló kihívásokat rejt magában, mint a közösségi hálózatok tanulmányozása. A „nagy adat”-ra jellemző, hogy mindig több szinten értelmezhető, és a méretétől függően újabb összefüggéseket, törvényszerűségeket tár elénk, így alkalmas lehet bizonyos dolgok előrejelzésére is.
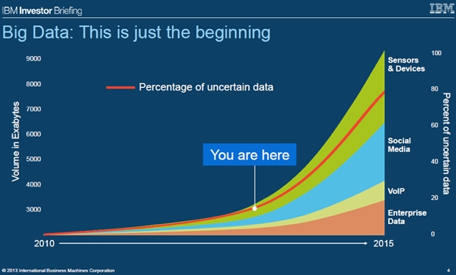
1. ábra: A “nagy adat” mennyisége várhatóan továbbra is exponenciális növekedést mutat majd, és az adatok hozzávetőleg 80%-ának kezelése láthatóan különleges módszereket igényel (http://www.sec.gov/Archives/edgar/data/51143/000110465913015636/a13-6155_18k.htm).
A csillagászat, például a Hubble űrtávcső által létrehozott nagyfelbontású képek hasonló informatikai kihívást jelentenek, mint a jelenleg a genomika alapját jelentő új generációs szekvenálás (DNS bázissorrend meghatározás) során készített képek. Ez utóbbi képek többszáz millió rövid (50-100 bázisos) DNS szekvencia leolvasását teszik lehetővé párhuzamosan egyszerre, a korábbi kisszámú hosszú (körülbelül 1000 bázisos) szekvencia leolvasásával szemben (http://en.wikipedia.org/wiki/DNA_sequencing). Míg az ember teljes genetikai információjának, a humán genomnak a meghatározásához először egy évtized sem volt elegendő (1990-2003) (http://en.wikipedia.org/wiki/Human_Genome_Project), jelenleg néhány szakember közreműködésével (bár még nem túl olcsón) hetek alatt összerakható bárki teljes genetikai kódja. Az új generációs szekvenálás megjelenése robbanásszerű fejlődést hozott az összes nukleinsav (DNS/RNS) alapú módszer számára: 2008 óta sorra jelennek meg az újabbnál újabb nukleinsav omikai módszerek, amelyek elhozták a biológia- és orvostudományok egyik legújabb forradalmát. A humán genom meghatározása óta több tízezer személy genomja (többtíz petabájtnyi adat) lett meghatározva (2. ábra; http://ark-invest.com/genomic-revolution/declining-costs-of-genome-sequencing, http://en.wikipedia.org/wiki/1000_Genomes _Project, http://www.genomicsengland.co.uk/the-100000-genomes-project), de ez önmagában sajnos nem volt elegendő a várt robbanásszerű fejlődéshez az összetett betegségek okainak felismerésében és e betegségek orvoslásában. Kiderült, hogy a génállomány sokkal nagyobb részének van feladata, tehát sokkal nehezebb megtalálni a „hibás tényező”-ket, mint azt korábban gondolták. A fehérjekódoló gének mellett jelentős szerepe van az újabban felfedezett nagyszámú, nagyrészt ismeretlen működésű „RNS gén”-eknek és a génekhez tartozó szabályozó régióknak. Ezek a régiók bármilyen távol elképzelhetőek az általuk szabályozott génektől a genomban; megtalálásuk és gén(ek)hez rendelésük a biológia jelenlegi nagy kihívásai közé tartozik.
A genom szabályozási egységeinek együttműködését a funkcionális genomika vizsgálja, amely magában foglalja a szabályozó régiókat kötő fehérjék vizsgálatát (cisztromika, epigenomika), az újonnan képződött és érett RNS-ek (transzkriptomika) és fehérjék vizsgálatát (proteomika), és ezek kölcsönhatásainak kutatását (interaktomika). A betegségek gyógyításában nagy szerepe van az egyéni genetikai különbségeknek. Ezek vizsgálatára szakosodott a perszonális genomika, és a családok génállományának vizsgálatát is hívhatjuk összehasonlító genomikának. Az emberek bizonyos reakciói a különböző táplálék összetevőkre, gyógyszerekre és mérgekre genomjuk ismeretében megjósolhatóak (lesznek). Az ezzel foglalkozó tudományágak rendre a táplálkozási, farmako- és toxikogenomika. Magukra a táplálékokra az „étel-omika” szakosodott (http://en.wikipedia.org/wiki/Omics). A genomika persze nemcsak az emberrel és modellállatokkal (egér, ecetmuslica, fonálféreg) foglalkozhat; az orvostudományok mellett egyre nagyobb szerepe van a biológia- és agrártudományokban is. A genomika a különböző fajok genetikai kódjának meghatározásával és összehasonlításával folyamatosan felülírja a fejlődéstan és rendszertan tudományait és a gének kialakulásáról alkotott képünket, mivel az újonnan „megszekvenált” fajok egyre pontosabban kirajzolják az evolúciós törzsfát és egyre több információval szolgálnak a rokon gének eredetéről és változatairól (ami még az orvostudományokban is jól jöhet!). Az Ensembl adatbázis (és genomböngésző) kezdetben az elérhető gerincesek (ember és főemlősök, egér és rágcsálók, ragadozók és patások, madarak, halak, stb.) genetikai információit gyűjtötte össze egy helyen (innen a neve is: ensembl = együtt; http://www.ensembl.org/info/about/species.html); folyamatosan bővül, és mára kiegészült az elérhető alacsonyabbrendű állatokkal, például rovarokkal, növényekkel, gombákkal és baktériumokkal is (http://ensemblgenomes.org/info/genomes).
A DNS molekulák nagy számban történő párhuzamos szekvenálása egy egészen újszerű tudomány létrejöttét is lehetővé tette. A metagenomika különböző környezeti minták kevert génállományainak darabkáit vizsgálja. Ilyen környezeti minta lehet például a talaj, a vizek vagy a bőr- és bélflóra. Craig Venter kutatócsoportja földkörüli „óceánminta” szekvenálásából csak a Sargasso-tengerből 1.2 millió, korábban ismeretlen gént írt le, melyek legalább 1800 baktériumfajból származnak (http://www.jcvi.org/cms/research/projects/gos/overview, http://www.ncbi.nlm.nih.gov/pubmed/15001713). Az újonnan felfedezett gének a biológiai jelentőségük mellett ipari jelentőséggel is bírhatnak: A gének által kódolt enzimek különleges vegyületek létrejöttét segíthetik elő, amelyek hasznosak lehetnek például a gyógyszeripar számára. Az újonnan felfedezett enzimek hatékonyan bonthatják a szintetikus, ökológiai veszélyt jelentő, nehezen bomló vegyületeket; a hőtűrő bontóenzimek pedig például mosóporok összetevőivé válhatnak. Az emberi flóraelemek arányának megváltozása sok (például autoimmun) betegség kialakulásában szerepet játszik, ezek vizsgálata és akár transzplantációja egyre elfogadottabb, egyre több helyen általános orvosi gyakorlat (http://en.wikipedia.org/wiki/Fecal_bacteriotherapy, http://www.ncbi.nlm.nih.gov/pubmed/19963349).
A nukleinsavak tömeges meghatározása mellett kifejlesztették az adott sejt- vagy szövettípusra jellemző kis szerves molekulák (metabolomika), a zsíroldékony szerves molekulák (lipidomika) és a fehérjék összességének (proteomika) meghatározását is (http://en.wikipedia.org/wiki/Omics). Létezik olyan módszer is, amely megkülönbözteti a fehérjemódosításokat, még több alapot nyújtva a sejtek anyagcsere és jelátviteli útvonalainak és azok elváltozásainak tanulmányozásához (http://en.wikipedia.org/wiki/Signal_transduction). Az útvonalak hálózata (mellyel a rendszerbiológia vagy sziszteomika foglalkozik) bizonyos szereplők vagy kapcsolatok ismeretének hiányában és a kölcsönhatások sokfélesége miatt még nagyrészt feltáratlan (http://en.wikipedia.org/wiki/Systems_biology). A fő útvonalak bizonyos sejttípusokból ismertek, de mivel nincs két egyforma sejtünk, a hálózat nagyon eltérően működhet. A közvetlen kölcsönhatások „felszínei" ezért is nagyon fontosak, kutatásuk megköveteli a fehérjék felépítésének és dinamikájának részletekbe menő ismeretét. A szerkezeti bioinformatika nemcsak lineáris nukleinsav és aminosav szekvenciákkal, hanem a molekulák és komplexeik háromdimenziós szerkezetével is foglalkozik, például kis-RNS molekulák, enzimek vagy gyógyszermolekulák célpontjait kutatva.
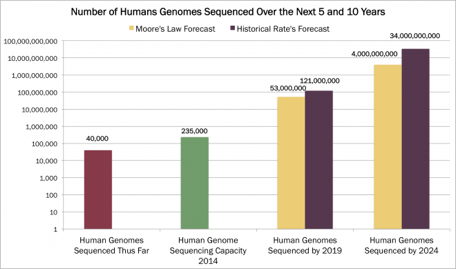
2. ábra: A jelenleg meghatározott, a 2014-ben meghatározható és a következő 5 illetve 10 évben várható humán genomok száma (http://ark-invest.com/genomic-revolution/declining-costs-of-genome-sequencing).
Az omikák adatainak és az egyéb „nagy adat”-oknak a kezelése és összehasonlítása általában több tudományterületet is érint. A molekuláris biológiában és genomikában használt műszerek nagy részének fejlesztését a biológusok mellett fizikusok, vegyészek, mérnökök és informatikusok végzik, de más területen is jellemző, hogy az adatok begyűjtésének és feldolgozásának a különböző stádiumai különböző szaktudást igényelnek. A „nagy adat”-ok kezeléséhez minden esetben szükség van az informatikában jártas szakemberre. A Gartner (amerikai információtechnológiai vállalat) előrejelzése szerint a „nagy adat” 2015-ben világszerte 4.4 millió új munkahelyet fog teremteni, melynek valószínűleg a kétharmada betöltetlen marad (1. ábra; http://hvg.hu/tudomany/20140513_3_millio_szabad_allas_lesz_bigdata). Az új adatok jelentős részét az emberi genom által kódolt információ adhatja, ugyanis az előrejelzések azt mutatják, hogy tíz éven belül az emberek nagyobb fele (több mint 4 milliárd ember!) rendelkezhet a saját genetikai kódjával (2. ábra). Emellett persze ott lesznek a kísérleti, diagnosztikai és más fajokból származó szekvencia- és egyéb információk is. Kérdéses viszont, hogy a „nagy adat”-nak köszönhető állásokból mennyit tudnak végül betölteni. A bioinformatikusok többsége biológusból képezte át magát, így nem váltak programozóvá, de együtt tudnak dolgozni az informatikusokkal és akár önálló informatikai fejlesztésre is képesek lehetnek. Egyre inkább jellemzővé válik ez a fajta átképzés és együttműködés a különböző területről érkezett szakemberek között, de ennek oktatása Európában a legtöbb helyen sajnos nem eléggé hatékony. A fogyasztói igények azonban előbb-utóbb ezen a téren is el kell, hogy érjék az elvárt fejlődést, így remélhetőleg hamarosan, az adatmennyiség rohamos növekedésének ellenére is csökkenni fog a szakemberhiány mértéke.

A kutatás a TÁMOP 4.2.4.A/2-11-1-2012-0001 azonosító számú Nemzeti Kiválóság Program – Hazai hallgatói, illetve kutatói személyi támogatást biztosító rendszer kidolgozása és működtetése országos program című kiemelt projekt keretében zajlott. A projekt az Európai Unió támogatásával, az Európai Szociális Alap társfinanszírozásával valósul meg.


Utolsó kommentek